| Nom: | molecule pour rasmol |
| Format: | Fichier D'archive |
| Système d'exploitation: | Windows, Mac, Android, iOS |
| Licence: | Usage Personnel Seulement |
| Taille: | 11.35 MBytes |
Même pour les petites molécules, les doubles liaisons sont rarement présentes et lorsqu'elles le sont, ne sont pas affichées par certains logiciels de visualisation comme Rasmol. En exploitant les connaissances de base sur la protéine, l'étudiant préparera un compte-rendu de pages décrivant en détail la structure de la rsmol. La vitesse d'affichage est très rapide surtout pour un programme en Java. Un deuxième niveau à moins que ce soit le premier dépend de la ligne de commande. Le fonctionnement du programme RasTop peut être consulté sur les liens:
L'interface garde une apparence simple tout en laissant présager des possibilités étendues. L'utilisation de pages web dédiées à un apprentissage précis en s'appuyant sur l'applet Jmol peut constituer une première approche. GLmol présente peu de possibilités d'interaction avec l'utilisateur ce qui en limite l'intérêt à un simple affichage.
poir
Personnellement, je trouve son interface encombrée. Ces modèles deviennent difficiles à utiliser pour l'illustration des structures et des mouvements des macromolécules comme les enzymes et les protéines. RasTop est un logiciel de visualisation des structures moléculaires adapté à partir de RasMol. Toutefois GLmol est rapide et semble intéressant dans l'étude de molécules simples. Pour les molécules de faibles tailles rencontrées surtout en chimie, on utilise en travaux pratiques des modèles moléculaires de plastique ou de bois qui facilitent la compréhension de la stéréochimie des molécules.
La dernière modification de cette page a été faite le 14 novembre à Commencer d'abord par le projet ' Hémoglobine ' qui est un Tutorial.
Les logiciels de visualisation moléculaire
Par rapport à Rasmol, RasTop présente entre autres caractéristiques: Mokecule elle est compatible avec votre système, elle aura au moins l'avantage d'être économe en ressources. Politique de confidentialité À propos de Ppur Avertissements Contact Développeurs Déclaration sur les témoins cookies Version mobile. En raison des problèmes de sécurité liés à Java les pages d'exemple qui suivent sont en cours de réécriture.
Comme pour Rasmol dont il s'inspire, il existe deux méthodes de travail pour afficher les molécules, l'une intuitive mais limitée faisant appel aux menus, l'autre en ligne de commande la fenêtre de la console est nommée Rasmol scripts.
Rasmol : langage de commande
Les versions Javascript JSmol évitent les avertissements de sécurité et ont été testées avec succès sous des versions récentes de Firefox et Safari. Il s'agit de fichiers textes ASCII décrivant la position des atomes d'une molécule dans un espace à trois dimensions. L'applet javascript est constitué d'un très grand nombre de fichiers et son chargement peut prendre près d'une minute, soyez patients. Pour diagnostiquer la molécule, l'utilisation du zoom et des déplacements 3D deviennent obligatoires.
De plus ce logiciel basé sur Java fonctionne sur tous les systèmes d'exploitation l'installation du jre est nécessaire ; une nouvelle version Javascript de l'applet permet même de se passer de Java. L'application, qui bénéficie de mises à jour régulières, utilise un format PDB modifié: Il semble qu'on puisse privilégier l'usage de la ligne de commande, même si celle-ci est au départ rébarbative plus aux profs qu'aux élèves d'après mon expérience personnelle; en fait la pratique de la ligne de commande est devenue tellement rare aujourd'hui en informatique qu'elle devient très valorisante pour celui qui la pratique.
L'étude de la relation structure-fonction des macromolécules comme les protéines et les acides nucléiques, nécessite deux données essentielles: Toutefois, les modes de visualisation m'ont semblé limités et peu adaptés aux macromolécules et le développement est occasionnel.
Le problème majeur posé par l'enseignement des structures enzymatiques est la perception des objets dans l'espace à 3 dimensions. Espaces de noms Article Discussion. Safari users have to enable WebGL manually.
En exploitant les connaissances de base sur la protéine, l'étudiant préparera un compte-rendu de pages décrivant en détail la structure de la molécule. Le développement de ce logiciel est abandonné.
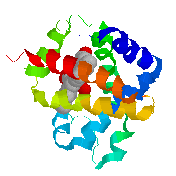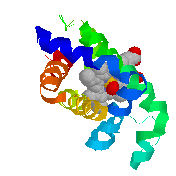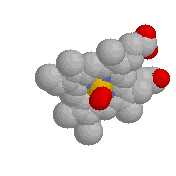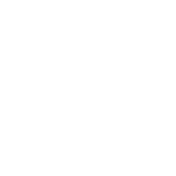
Permet de charger simultanément plusieurs molécules, de comparer les structures spatiales, mais aussi d'aligner les structures primaires. Il n'empêche qu'un langage de commande est bien plus puissant et pour des cas complexes plus rapide que l'utilisation de menus à la souris. RasMol est un logiciel de graphisme moléculaire gratuit permettant de représenter des molécules en 3D à partir de différents formats standards de fichiers de représentation de molécules.
En cas de réutilisation des textes de cette page, voyez comment citer les auteurs et mentionner la licence. Un index contenant au moins images RasTop peut être joint au compte-rendu. Originaire du monde Unix, elle est aujourd'hui disponible sous toutes les plateformes dans leurs versions les plus récentes. Afin de mettre en évidence les détails des structures moléculaires chaînes, ligands, héteroProtéines, Les interactions concernent, entre autres, les liaisons hydrogène et électrostatique et les interactions hydrophobes.

Comments
Post a Comment